Next-Generation-Sequenzierung – Panels, Library-Preparation-Kits und Software für die Zytogenetik und hämatologische Forschung
SureSeq umfasst ein ständig wachsendes Portfolio von NGS-Panels für die Forschung im Bereich hämatologischer Tumore sowie Library Preparation Kits für den genauen Nachweis einer Vielzahl genetischer Aberrationen.
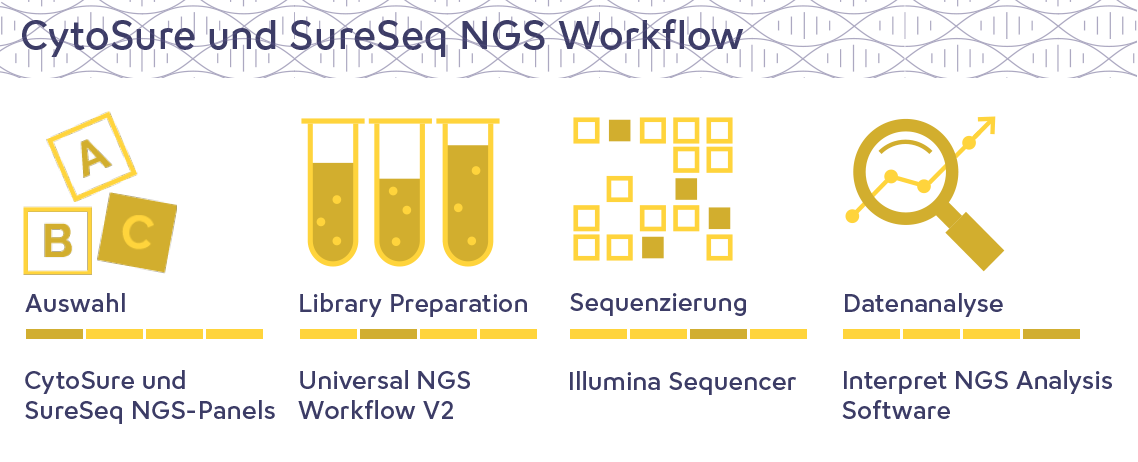
SureSeqTM NGS Panels – Leistungsstarke Next Generation Sequencing Lösungen für die hämatologische Forschung
- Jede Information zählt. Daher entsprechen SureSeq Panels immer dem aktuellen Stand der Forschung.
- Detektieren Sie auch niedrigfrequente SNVs, Indels, ITDs, PTDs, CNVs, LOH und Translokationen mit hoher Sicherheit.
- Die SureSeq Hybridisierungs-Panels zeichnen sich durch eine hohe Lesetiefe und eine sehr gleichmäßige und gute Abdeckung aus.
- Für eine effiziente Auswertung und klare Darstellung der Sequenzierungsdaten werden alle SureSeq-Panels mit unserer leistungsstarken und anwenderfreundlichen Interpret Analysesoftware geliefert.
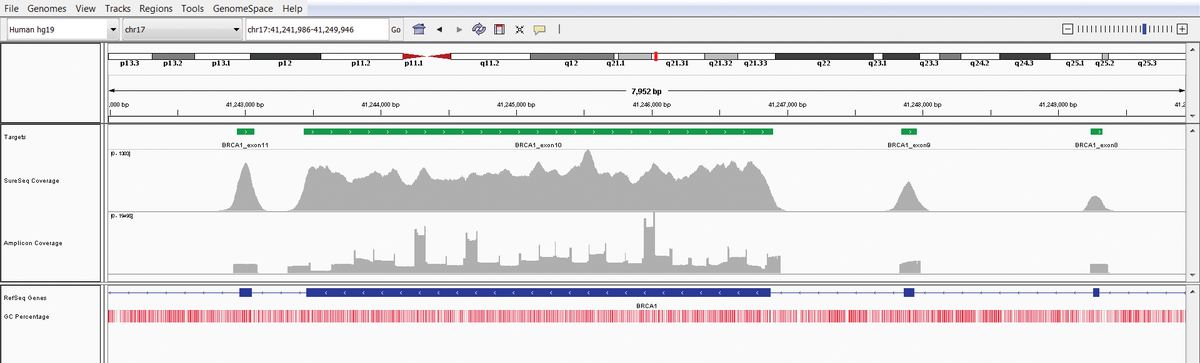
Für die wichtigsten Erkrankungen im Bereich hämatologischer Neoplasien wie CLL oder AML gibt es vorkonfektionierte Panels, die sich in gewissem Rahmen für Sie auch modifizieren lassen. Mit einem SureSeq myPanel stellen wir das exakt auf Ihre Fragestellung zugeschnittene Panel mit den Experten von OGT individuell für Sie zusammen.
Folgende SureSeq-Katalog-Panels sind aktuell erhältlich. Jedes dieser Panels kann unproblematisch auf Ihre Bedürfnisse zugeschnitten werden:
| Panelgröße | Zielgene | Abberationen | Mittlere Zielabdeckung | |
| Myeloid fusion Panel | 61 kb. | 30 gene fusions | Fusion, MECOM expression | Ca. 1000x (VAF 1 %) |
| CLL + CNV Panel v3 | 142 kb. | 16 | SNV, indel, CNV | > 700x (VAF 1 %) |
| Myeloid-MRD Panel | 11,2 kb. | 13 | SNV, indel, ITD | Bis 20.000x (VAF 0,05 %) |
| Myeloid Plus Panel | 132 kb. | 49 | SNV, indel, ITD, PTD | > 700x (VAF 2-5 %) |
| CLL + CNV Panel v2 | 117 kb. | 13 | SNV, indel, CNV | > 1000x (VAF 1 %) |
| Core MPN Panel | 1 kb. | 3 | SNV, indel | > 1000x (VAF 1 %) |
| Pan-Myeloid Panel | 221 kb. | 70 | SNV, indel, ITD, PTD | > 1000x (VAF 1 %) |
| Germline Breast Cancer + CNV | 52,6 kb. | 7 | SNV, indel, CNV | > 250x |
| Core MPN Panel + BLC-ABL | 22 kb. | 4 | SNV, indel, fusion | > 1000x (VAF 1 %) |
Next Generation Sequencing heute
In den letzten 20 Jahren hat das Next Generation Sequencing (NGS) im klinischen Forschungslabor eine rasche Verbreitung gefunden, da es im Vergleich zu herkömmlichen Methoden mehrere Gene oder Genregionen gleichzeitig in einem einzigen Test analysieren kann. [1]
Während die Erkennung von Einzel-Nukleotid-Varianten sowie kleinen Deletionen und Insertionen in Panel-Sequenzierungsdaten unproblematisch ist, stellt die Identifizierung von Rearrangements wie Multi-Exon Copy Number Variations (CNVs) immer noch eine große Herausforderung dar. Keimbahn-CNVs sind bekanntlich eine Quelle der genetischen Vielfalt beim Menschen, aber auch die Ursache verschiedener Erbkrankheiten, die im Zusammenhang mit einer Vielzahl von mendelschen und somatischen genetischen Störungen stehen. Übliche Methoden zum Nachweis von CNVs sind die Multiplex Ligation-dependent Probe Amplification (MLPA), die Array-Comparative Genomic Hybridization (aCGH) und die qPCR. Diese Methoden haben jedoch viele Nachteile, z. B. sind sie komplex und kostspielig und erfordern Vorkenntnisse der zu analysierenden Region. [2]
Hochdurchsatztechnologien wie NGS begründeten das Zeitalter der Big Data in Biologie und Medizin. Die Menge der zu verarbeitenden Daten kann für Forscherinnen und Forscher ohne Bioinformatik-Know-how überwältigend sein. Daher ist die Entwicklung zuverlässiger, reproduzierbarer und benutzerfreundlicher Softwaretools unerlässlich, um aus den NGS-Daten Informationen über molekulare Merkmale abzuleiten. [3]

SureSeq NGS-Panels – Flexibel, zuverlässig und auf Ihre Forschung abgestimmt
SureSeqTM NGS-Panels wurden in Zusammenarbeit mit anerkannten Krebsexpertinnen und -experten entwickelt, um wichtige Aberrationen zu erkennen, die bei einer Vielzahl von hämatologischen Krebsarten auftreten. SureSeq myPanel bieten Ihnen die Flexibilität, die im Katalog verfügbaren Panels einfach an Ihre Forschungsanforderungen anzupassen. Kombinieren Sie einfach die Gene, exonischen und intronischen Inhalte, die Sie benötigen, um das NGS-Krebspanel zu erstellen, das genau Ihren Anforderungen entspricht.
Das einzigartige Design des SureSeq-Panels in Verbindung mit der hybridisierungsbasierten Anreicherung bietet eine vollständige und einheitliche Abdeckung. Es ermöglicht die genaue Erkennung von SNVs und Indels mit niedriger Frequenz sowie von strukturellen Aberrationen wie ITDs, PTDs, CNVs, LOH und Translokationen. In Kombination mit unseren verschiedenen SureSeq-Library Preparation Kits erleichtert SureSeq die Durchführung mehrerer Assays und rationalisiert Ihre Forschung, indem es umfassende Ergebnisse mit einem einzigen NGS-Workflow liefert.
Alle SureSeq-Panels werden mit Interpret geliefert, einer leistungsstarken und benutzerfreundlichen NGS-Analyselösung, die eine mühelose Übersetzung all Ihrer NGS-Daten in aussagekräftige Ergebnisse ermöglicht. Wählen Sie zwischen einer lokalen Installation von Interpret oder Interpret on-Cloud. SureSeq-Panels können in Kombination mit der Interpret NGS-Analysesoftware ein breites Spektrum an Varianten und strukturellen Aberrationen erkennen.
| SNVs und Indels | Erkennung von SNVs und Indels mit niedriger Frequenz bis zu 1 % VAF (0,05 % VAF für MRD), einschließlich schwer zu sequenzierender Regionen (z. B. CEBPA) |
| ITDs | Erkennung von FLT3-ITDs von wenigen Basenpaaren bis hin zu >200 bp |
| PTDs | Erkennung von KMT2A-PTDs aller Größen |
CNVs und LOH | Erkennung von CNVs, die vom Verlust einzelner Exons bis zu ganzen Chromosomenarmen reichen, sowie Trisomien |
| Translokationen | Erkennung von BCR-ABL-Translokationen |
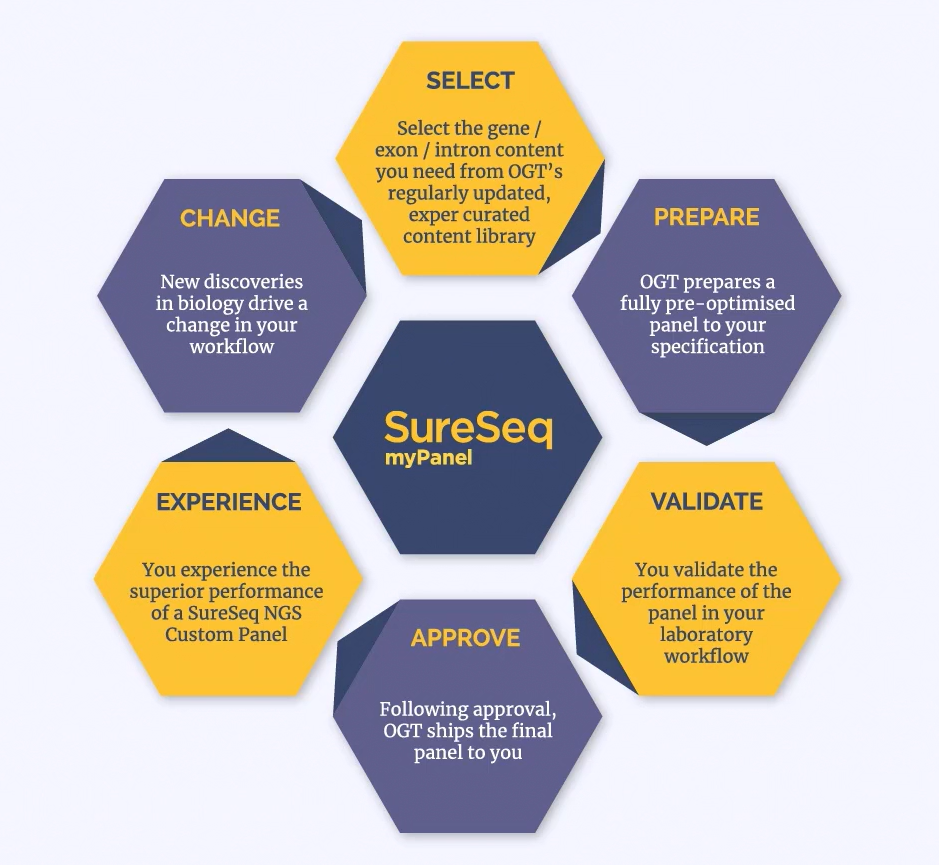
SureSeq myPanel – Ihr individualisierbares NGS-Krebspanel
Umfassend oder gezielt? Hotspots oder Exons?
Stellen Sie Ihr ideales NGS-Krebs-Panel individuell zusammen und konzentrieren Sie sich auf genau das, was für Ihre Forschung relevant ist. Werfen Sie einen Blick auf die voroptimierten Inhalte von SureSeq myPanel, um die Assay-Entwicklungszeit und den internen Optimierungsaufwand auf ein Minimum zu reduzieren.
Designen Sie Ihr individuelles SureSeq myPanel.
Referenzen
[1] Yohe, S., & Thyagarajan, B. (2017): Review of Clinical Next-Generation Sequencing. Archives of pathology & laboratory medicine, 141(11), 1544–1557.
[2] Sorrentino, E., Daja, M., Cristofoli, F., Paolacci, S., Bertelli, M., & Marceddu, G. (2021): CNV analysis in a diagnostic setting using target panel. European review for medical and pharmacological sciences, 25(1 Suppl), 7–13.
[3] Dotolo, S., Esposito Abate, R., Roma, C., Guido, D., Preziosi, A., Tropea, B., Palluzzi, F., Giacò, L., & Normanno, N. (2022): Bioinformatics: From NGS Data to Biological Complexity in Variant Detection and Oncological Clinical Practice. Biomedicines, 10(9), 2074.